Projet : Développement de la plateforme MEDomicsLab
État
En cours (2020-aujourd’hui)
Type
Général
Équipe
- Andréanne Allaire1 (2022-2024)
- Brahim Fakri1 (2022-2025)
- Cedrik Lampron1 (2022-2025)
- Charles Lévesque-Matte1 (automne 2020)
- Charles-Olivier Ipperciel1 (2024-aujourd’hui)
- Clarisse Cheng1 (été 2022)
- Corentin Gauthier1 (été 2022)
- Guillaume Blain1 (été 2022-aujourd’hui)
- Hithem Lamri1 (automne 2022-2023)
- Jonathan Perron1 (2020-2021)
- Kayla Davio-Roy1 (été 2021)
- Ludmila Amriou1 (2023-2024)
- Lyna Hiba Chikouche1 (2023-2024)
- Mahdi Ait Lhaj Loutfi1 (2021-aujourd’hui)
- Mamadou Mountagha Bah1 (2020-2021)
- Mariem Kallel1 (2024-aujourd’hui)
- Mohammed Benabbassi1 (automne 2022)
- Nicolas Longchamps1 (été 2022-aujourd’hui)
- Ouael Nedjem Eddine SAHBI1 (2023-aujourd’hui)
- Robin Mailhot1 (automne 2020)
- Sarah Denis1 (2023-2024)
- Martin Vallières1 (2020-aujourd’hui)
1 Département d’informatique, Université de Sherbrooke, Sherbrooke (QC), Canada
Description
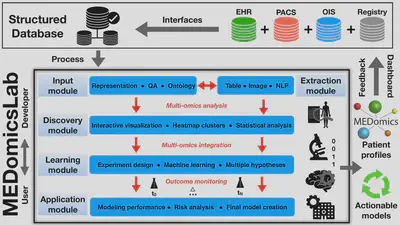
MEDomicsLab est une plateforme de calcul open-source pour la modélisation intégrative des données en médecine. Elle a été créée par un consortium international de scientifiques médicaux (https://medomics.ai), dont l’objectif principal est de faciliter le développement et la traduction clinique des applications de l’intelligence artificielle (IA) en médecine. La plateforme est basée sur le langage de programmation Python et contient cinq modules majeurs qui permettent de charger, traiter, explorer des données multi-omiques, et de créer et évaluer des modèles exploitables pour la médecine de précision. Le flux de travail intrinsèque de MEDomicsLab est conçu pour fournir différents niveaux d’abstraction de la complexité méthodologique aux utilisateurs et aux développeurs via des scripts d’application, des paramètres d’option et des structures de classe. En général, MEDomicsLab est envisagé pour être au centre de équipes de recherche multidisciplinaires et des infrastructures de bases de données hospitalières.
Les récents progrès des technologies dites « omiques » (génomiques, radiomiques, protéomiques, etc.) offrent des possibilités sans précédent pour caractériser les processus biologiques qui sont reliés aux phénotypes des maladies. La combinaison efficace de ces éléments en tant que « modélisation multi-omique » nous permettra certainement de mieux adapter les traitements à chaque patient (c.-à-d. « médecine de précision »)
Étant donnée la complexité des problèmes médicaux, l’atteinte du plein potentiel de la médecine de précision est directement reliée à notre capacité à bien gérer, structurer et tirer profit des bases de données de nos hôpitaux. Dès lors, la maîtrise et la combinaison des techniques d’analyse de données suivantes devraient augmenter l’impact des modèles de prédiction « multi-omiques » qui sont construits à partir de ces bases de données:
- Analyse d’image : l’extraction des caractéristiques radiomiques nous permet de mieux caractériser l’hétérogénéité tumorale. Le logiciel MEDimage est la composante consacrée pour cette tâche.
- Apprentissage automatique : certaines techniques d’apprentissage permettent de mieux combiner les données de différentes catégories (telles que multi-omiques).
- Apprentissage profond : la multitude d’architectures de réseaux de neurones profonds offre plusieurs possibilités par rapport à l’apprentissage automatique de différentes tâches en médecine. La performance de certains réseaux est parfois supérieure à celle des humains.
- Traitement automatique du texte : le traitement et le codage automatique des comptes rendus textuels des dossiers électroniques des patients permettraient une meilleure intégration d’informations pertinentes à la modélisation multi-omique durant toute la période de prise en charge des patients.
- Apprentissage fédéré : afin d’augmenter la quantité de données modélisées provenant de différents établissements de santé, une approche d’apprentissage distribué peut être utilisée - les données sont conservées dans les limites de chaque établissement, évitant ainsi les problèmes de transfert de données et de confidentialité.
Pour plus d’informations, voir le site web du projet.
Cette plateforme est développée en collaboration avec :
- University California San Francisco (Olivier Morin, Taman Upadhaya, Jorge Barrios)
- University of Toronto (Jan Seuntjens)
- The D-Lab (Philippe Lambin, Henry Woodruff, Avishek Chatterjee)
- Oncoray (Alex Zwanenburg, Steffen Löck)
- Université Laval (Louis Archambault, Philippe Després)